遺傳進化算法進行高效特征選擇
在構建機器學習模型時,特征選擇是一個關鍵的預處理步驟。使用全部特征往往會導致過擬合、增加計算復雜度等問題。因此,我們需要從原始特征集中選擇一個最優子集,以提高模型的泛化性能和效率。
特征選擇的目標是找到一個二元掩碼向量,對應每個特征的保留(1)或剔除(0)。例如,對于10個特征,這個掩碼向量可能是[1,0,1,1,0,0,1,0,1,0]。我們需要通過某種優化方法,尋找一個使目標函數(如模型的貝葉斯信息準則BIC)最小化的最優掩碼。
當特征數量較小時,我們可以使用暴力搜索等枚舉方法。但隨著特征數量的增加,搜索空間將呈指數級增長,枚舉搜索將變得無法承受。這時我們需要借助啟發式優化算法,例如遺傳算法、模擬退火等,在有限時間內找到一個近似最優解。
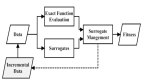
本文以Kaggle房價數據集(213個特征)為例,比較了經典的序貫特征選擇算法(SFS)和一種稱為進化策略(CMA-ES)的啟發式優化算法在特征選擇任務上的表現。實驗結果表明,CMA-ES相比SFS能更快地converge到更優的特征子集,從而獲得更小的BIC值和更佳的模型泛化性能
GA - 遺傳算法
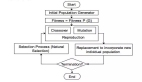
遺傳算法是一種啟發式優化算法,其靈感源自達爾文的進化論和自然選擇理論。在自然界中,生物個體通過基因遺傳,攜帶有利于生存和繁衍的特征,經過漫長的自然選擇過程,適者生存,不斷進化。類似地,遺傳算法也是通過模擬這一自然進化過程,對問題的潛在解決方案(個體)進行遺傳、變異和選擇,最終得到滿足目標條件的最優或近似最優解。
以特征選擇為例,假設我們有N個特征,需要確定一個長度為N的二進制向量[1, 0, 0, 1, 1, 1, ...]來表示選擇(1)或剔除(0)每個特征,目標是找到一個使成本函數或目標函數最小化的最優特征組合。我們可以將每個這樣的二進制向量看作一個"個體",每個位置上的0或1就是該個體的一個"基因"。
遺傳算法基本步驟:
- 評估:計算每個個體的適應度(目標函數值)。
- 選擇:根據適應度挑選出若干個體作為父代。
- 交叉:隨機選擇兩個父代個體,按一定交叉方式組合它們的基因,產生新的個體(子代)。
- 變異:以一定的小概率改變子代個體中的某些基因值。
- 替換:用新產生的子代個體替換種群中適應度較低的個體。
通過不斷迭代上述過程,種群中個體的平均適應度就會不斷提高,最終可以得到滿足要求的最優或近似最優解。
一些關鍵概念和組件:
- 個體(Individual):表示種群中的一個候選解,通常用染色體(一串數字或字符)來表示。
- 種群(Population):包含多個個體的集合。
- 適應度函數(Fitness Function):用于評估每個個體的優劣程度。
- 選擇(Selection):根據適應度函數的結果,從當前種群中選擇部分個體作為父代,用于產生下一代種群。
- 交叉(Crossover):將兩個父代個體的染色體進行組合,產生一個或多個新的子代個體。
- 變異(Mutation):通過改變個體染色體上的某些位,增加種群的多樣性。
- 最佳個體(Hall of Fame):用于保存迄今為止找到的最佳個體,防止在進化過程中被淘汰或丟失。
- 優秀個體(Elitism):在每一代中,直接將當前種群中的優秀個體傳遞到下一代,而不需要經過選擇、交叉和變異等操作。
遺傳算法首先隨機生成一組這樣的個體,構成初始種群。比如,如果N=12,種群數量為8,那么初始種群可能包含8個長度為12的隨機二進制向量。
 遺傳算法-種群
遺傳算法-種群
群體創建后,通過目標函數對每個個體進行評估。
接下來是選擇階段,旨在保留具有良好特征的個體,淘汰表現不佳的個體。這一過程有多種策略可供選擇,從簡單的排序法到更加復雜的隨機錦標賽選擇等。選擇策略需要權衡探索(嘗試新的解決方案)和利用(保留已知的良好解決方案)之間的平衡。遺傳算法的核心在于通過有效探索來尋找最優解。常見的選擇技術包括:錦標賽選擇、輪盤賭選擇、等級選擇等。
經過選擇后,遺傳算法會在保留的優秀個體基因庫中引入變異,以產生新的候選解。主要有兩種變異技術:交叉和突變。
交叉是模仿生物交配過程,將兩個親代個體的部分基因隨機組合,產生新的子代個體。這一過程能夠將親代的良好特征組合在一起,有望產生更優秀的后代。
突變則是對個體的基因做出少量隨機改變,以引入新的多樣性,避免群體過早收斂于局部最優解。
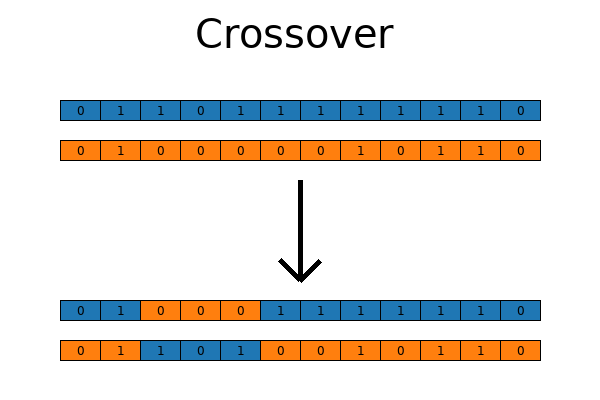 遺傳算法-交叉
遺傳算法-交叉
變異,同樣是指自然界中遺傳物質發生隨機突變,基因庫中引入新的價值,從而增加其多樣性。
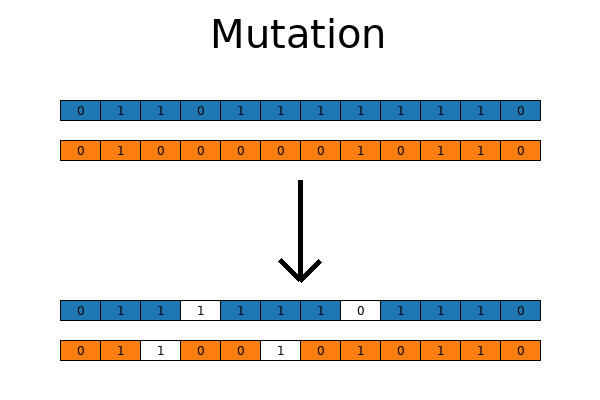 遺傳算法-變異
遺傳算法-變異
之后,算法循環往復:再次通過目標函數評估個體,進行選擇,然后是交叉、變異等。
可以使用各種停止標準:如果目標函數在一定代數內停止改進,循環就會中斷。或者你也可以設定一個硬性的停止條件,即已評估的總代數。或者以時間為基礎,或者觀察外部信號等。無論如何,具有最佳目標值的個體都應被視為問題的解決方案。
在遺傳算法(GA)中,隨機選擇技術有時可能會由于偶然因素而放棄當代種群中最優秀的個體。盡管發生概率不高,但這種情況確實存在。這就是引入"精英主義"策略的目的,它確保無論如何,種群中的優秀個體都會被保留下來。然而,過度使用精英主義也可能導致算法陷入局部最優解,從而錯失全局最優解。GA本質上是一種探索性算法,根據我有限的經驗,為其引入過多偏見并不利于尋找最優解。GA本身就提供了大量的算法變體可供嘗試和探索。
在GA中,我們可以調整以下幾個超參數:
- 種群規模(個體數量)
- 基因突變概率
- 個體交叉概率
- 個體選擇策略等
手動嘗試不同超參數組合是尋找最優參數的一種方式。另一種高級方法是將GA封裝到優化框架(如Optuna)中,利用框架自動搜索最佳超參數,但這種方式計算代價較高。
無論采用何種方式,GA都為我們提供了一種靈活有趣的優化探索方式,讓我們可以充分發揮創意。
特征選擇的GA代碼
下面是一個可用于特征選擇的簡單 GA 代碼。它使用了功能強大的 deap 庫,但學習曲線可能比較陡峭。不過,這個簡單的版本應該足夠清晰。
上下滑動查看更多源碼# to maximize the objective
# fitness_weights = 1.0
# to minimize the objective
fitness_weights = -1.0
# copy the original dataframes into local copies, once
X_ga = X.copy()
y_ga = y.copy()
# 'const' (the first column) is not an actual feature, do not include it
X_features = X_ga.columns.to_list()[1:]
try:
del creator.FitnessMax
del creator.Individual
except Exception as e:
pass
creator.create("FitnessMax", base.Fitness, weights=(fitness_weights,))
creator.create(
"Individual", array.array, typecode='b', fitness=creator.FitnessMax
)
try:
del toolbox
except Exception as e:
pass
toolbox = base.Toolbox()
# Attribute generator
toolbox.register("attr_bool", random.randint, 0, 1)
# Structure initializers
toolbox.register(
"individual",
tools.initRepeat,
creator.Individual,
toolbox.attr_bool,
len(X_features),
)
toolbox.register("population", tools.initRepeat, list, toolbox.individual)
def evalOneMax(individual):
# objective function
# create True/False selector list for features
# and add True at the start for 'const'
cols_select = [True] + [i == 1 for i in list(individual)]
# fit model using the features selected from the individual
lin_mod = sm.OLS(y_ga, X_ga.loc[:, cols_select], hascnotallow=True).fit()
return (lin_mod.bic,)
toolbox.register("evaluate", evalOneMax)
toolbox.register("mate", tools.cxTwoPoint)
toolbox.register("mutate", tools.mutFlipBit, indpb=0.05)
toolbox.register("select", tools.selTournament, tournsize=3)
random.seed(0)
pop = toolbox.population(n=300)
hof = tools.HallOfFame(1)
pop, log = algorithms.eaSimple(
pop, toolbox, cxpb=0.5, mutpb=0.2, ngen=10, halloffame=hof, verbose=True
)
best_individual_ga_small = list(hof[0])
best_features_ga_small = [
X_features[i] for i, val in enumerate(best_individual_ga_small) if val == 1
]
best_objective_ga_small = (
sm.OLS(y_ga, X_ga[['const'] + best_features_ga_small], hascnotallow=True)
.fit()
.bic
)
print(f'best objective: {best_objective_ga_small}')
print(f'best features: {best_features_ga_small}')代碼使用對象來定義個體和整個種群,并采取評估(目標函數)、交叉/交配、突變和選擇的策略。起初,代碼從包含 300 個個體的種群開始,然后調用 eaSimple() 函數(該函數包含了交叉、變異和選擇的預設序列),并且僅運行 10 代以簡化過程。名人堂的規模為 1,它會保留絕對最優秀的個體,以防它們在選擇等過程中被意外變異或跳過。
名人堂并非精英主義,而是從群體中復制最優秀的個體,并僅在錫罐中保留一個非活動副本。精英主義則是將活躍種群中最優秀的個體一代一代地保留下來。
這個簡單的代碼很容易理解,但效率很低。下面有更復雜版本的 GA 代碼,更復雜、更優化的代碼 1000 代后。
# to maximize the objective
# fitness_weights = 1.0
# to minimize the objective
fitness_weights = -1.0
# copy the original dataframes into local copies, once
X_ga = X.astype(float).copy()
y_ga = y.astype(float).copy()
# 'const' (the first column) is not an actual feature, do not include it
X_features = X_ga.columns.to_list()[1:]
try:
del creator.FitnessMax
del creator.Individual
except Exception as e:
pass
creator.create("FitnessMax", base.Fitness, weights=(fitness_weights,))
creator.create(
"Individual", array.array, typecode='b', fitness=creator.FitnessMax
)
try:
del toolbox
except Exception as e:
pass
toolbox = base.Toolbox()
# Attribute generator
toolbox.register("attr_bool", random.randint, 0, 1)
# Structure initializers
toolbox.register(
"individual",
tools.initRepeat,
creator.Individual,
toolbox.attr_bool,
len(X_features),
)
toolbox.register("population", tools.initRepeat, list, toolbox.individual)
def evalMany(individuals):
# individuals is an array of shape (n_individuals, n_features)
# transform it into a list of lists:
# - a list of individuals
# - each individual is a list of feature selectors
ind_list = [list(i) for i in list(individuals)]
ret = []
for ind in ind_list:
# create a list of True/False feature selectors from each individual
# pre-pend True to always select the 'const' feature
cols_select = [True] + [i == 1 for i in list(ind)]
# fit model using the features selected from the individual
lin_mod = sm.OLS(y_ga, X_ga.loc[:, cols_select], hascnotallow=True).fit()
ret.append((lin_mod.bic,))
return ret
# multiprocess pool to evaluate individuals
def joblib_map(f, njobs, *iters):
return Parallel(n_jobs=njobs)(delayed(f)(*args) for args in zip(*iters))
def selElitistAndTournament(
individuals, k_tournament, k_elitist=0, tournsize=3
):
# elitist tournament
# in addition to the regular tournament, ensure the top #k_elistist individuals are preserved
return tools.selBest(individuals, k_elitist) + tools.selTournament(
individuals, k_tournament, tournsize
)
# Hyperparameters
population_size = 1000
crossover_probability = 0.5
individual_mutation_probability = 0.2
gene_mutation_probability = 0.1
tournament_size = 3
elite_size = 0
n_jobs = os.cpu_count()
# register the pool as the mapper
# and the custom function as the evaluator
toolbox.register("map_multi", joblib_map)
toolbox.register("evaluate", evalMany)
toolbox.register("mate", tools.cxTwoPoint)
toolbox.register("mutate", tools.mutFlipBit, indpb=gene_mutation_probability)
# toolbox.register("select", tools.selTournament, tournsize=tournament_size)
# selection with tournament and optional elitism
toolbox.register(
"select",
selElitistAndTournament,
k_tournament=population_size - elite_size,
k_elitist=elite_size,
tournsize=tournament_size,
)
random.seed(0)
population = toolbox.population(n=population_size)
hall_of_fame = tools.HallOfFame(1)
objective_runs_ga = 0
# Evaluate the individuals with an invalid fitness
invalid_ind = [ind for ind in population if not ind.fitness.valid]
# split the population in a list with n_jobs elements
# each element is an array containing multiple individuals
# send individuals to the evaluator
fitnesses_nested = toolbox.map_multi(
toolbox.evaluate, n_jobs, np.array_split(invalid_ind, n_jobs)
)
objective_runs_ga += len(invalid_ind)
fitnesses = []
for l in fitnesses_nested:
fitnesses += l
for ind, fit in zip(invalid_ind, fitnesses):
ind.fitness.values = fit
hall_of_fame.update(population)
n_gen = 1000
best_objective_per_gen_ga = np.full(n_gen, np.nan)
best_objective_ga = np.nan
best_generation_ga = 0
gene_values_mean = np.zeros((n_gen, len(X_features)))
gene_maes = np.full(n_gen, np.nan)
time_to_best_ga = np.inf
# Begin the generational process
iterator = tqdm(range(1, n_gen + 1), desc='generation')
t_start = time.time()
for gen in iterator:
t_start_loop = time.time()
# Select the next generation of individuals
offspring = toolbox.select(population)
# Vary the pool of individuals via cross-over and mutation
offspring = algorithms.varAnd(
offspring,
toolbox,
crossover_probability,
individual_mutation_probability,
)
# Evaluate the individuals with an invalid fitness
invalid_ind = [ind for ind in offspring if not ind.fitness.valid]
# split the population in a list with n_jobs elements
# each element is an array containing multiple individuals
# send list to the evaluator pool
fitnesses_nested = toolbox.map_multi(
toolbox.evaluate, n_jobs, np.array_split(invalid_ind, n_jobs)
)
objective_runs_ga += len(invalid_ind)
fitnesses = []
for l in fitnesses_nested:
fitnesses += l
for ind, fit in zip(invalid_ind, fitnesses):
ind.fitness.values = fit
# Update the hall of fame with the generated individuals
hall_of_fame.update(offspring)
# Replace the current population by the offspring
population[:] = offspring
t_end_loop = time.time()
# record the mean gene values across the population
gene_values_mean[gen - 1, :] = np.array(population).mean(axis=0)
if gen >= 2:
gene_maes[gen - 1] = mean_absolute_error(
gene_values_mean[gen - 2, :], gene_values_mean[gen - 1, :]
)
# pick best individual for stats recording
best_individual_ga = tools.selBest(population, 1)[0]
best_objective_per_gen_ga[gen - 1] = best_individual_ga.fitness.values[0]
if (
best_objective_ga is np.nan
or fitness_weights * best_individual_ga.fitness.values[0]
> fitness_weights * best_objective_ga
):
best_objective_ga = best_individual_ga.fitness.values[0]
best_generation_ga = gen
time_to_best_ga = t_end_loop - t_start
print(
f'gen: {gen:4n}, curr/prev gene MAE: {gene_maes[gen - 1]:.4f}, new best objective: {best_objective_ga:.4f}, time to best: {time_to_best_ga:.4f}'
)
if os.path.isfile('break'):
# to gracefully break the loop, manually create a file called 'break'
print(f'Found break file, stopping now.')
iterator.close()
break
g_completed_ga = gen
best_individual_ga = list(hall_of_fame[0])
best_features_ga = [
X_features[i] for i, val in enumerate(best_individual_ga) if val == 1
]
best_objective_ga = (
sm.OLS(y_ga, X_ga[['const'] + best_features_ga], hascnotallow=True).fit().bic
)
## 強烈建議關注#公眾號:數據STUDIO 更多優質內容每日更新!best objective: 33705.569572544795
best generation: 787
objective runs: 600525
time to best: 158.027 sec同樣,在選擇任何特征之前的基準 BIC 計算:
上下滑動查看更多源碼print(f'best objective: {best_objective_ga}')
print(f'best generation: {best_generation_ga}')
print(f'objective runs: {objective_runs_ga}')
print(f'time to best: {time_to_best_ga:.3f} sec')
print(f'best features: {best_features_ga}')
gvm_df = (
pd.DataFrame(
gene_values_mean,
columns=X_features,
index=range(1, gene_values_mean.shape[0] + 1),
)
.sort_index(axis=1)
.iloc[:g_completed_ga]
)
if g_completed_ga > 20:
x_ticks = list(range(1, g_completed_ga + 1, round(g_completed_ga / 20)))
else:
x_ticks = list(range(1, g_completed_ga + 1))
if x_ticks[-1] != g_completed_ga:
x_ticks.append(g_completed_ga)
fig, ax = plt.subplots(
3,
1,
sharex=True,
height_ratios=[24, 1, 1],
figsize=(12, 48),
layout='constrained',
)
sns.heatmap(
gvm_df.sort_index(axis=1).T,
vmin=0.0,
vmax=1.0,
cmap='viridis',
cbar=True,
cbar_kws={'fraction': 0.01, 'anchor': (0.0, 1.0)},
ax=ax[0],
)
ax[0].set_title('Population average of gene values after each generation')
ax[0].axvline(x=best_generation_ga, color='C7')
ax[0].set_xlabel('generation')
ax[0].tick_params(axis='both', reset=True)
ax[0].set_xticks(x_ticks)
ax[0].set_xticklabels(x_ticks)
ax[1].set_xlabel('generation')
ax[1].plot(list(range(2, g_completed_ga + 1)), gene_maes[1:g_completed_ga])
ax[1].vlines(
x=best_generation_ga,
ymin=gene_maes[1:g_completed_ga].min(),
ymax=gene_maes[1:g_completed_ga].max(),
colors='C7',
)
ax[1].set_xlabel('generation')
ax[1].set_ylabel('MAE')
ax[1].set_title(
'Mean absolute error of average gene values between current generation and previous generation'
)
ax[1].tick_params(axis='both', reset=True)
ax[2].set_xlabel('generation')
ax[2].plot(
list(range(1, g_completed_ga + 1)),
best_objective_per_gen_ga[:g_completed_ga],
)
ax[2].vlines(
x=best_generation_ga,
ymin=min(best_objective_per_gen_ga[:g_completed_ga]),
ymax=max(best_objective_per_gen_ga[:g_completed_ga]),
colors='C7',
)
ax[2].set_xlabel('generation')
ax[2].set_ylabel('best objective')
ax[2].set_title('Best objective')
fig.suptitle('Genetic algorithm')
fig.savefig('ga-performance.png')
fig.show()best objective: 33705.569572544795
best generation: 787
objective runs: 600525
time to best: 158.027 sec
best features: ['BedroomAbvGr', 'BldgType_1Fam', 'BsmtFinSF1', 'BsmtFinSF2', 'BsmtUnfSF', 'Condition1_Norm', 'Condition2_PosN', 'ExterQual_Ex', 'Exterior1st_BrkFace', 'Functional_Sev', 'Functional_Typ', 'GarageArea', 'GrLivArea', 'HeatingQC_Ex', 'KitchenQual_Ex', 'LandContour_HLS', 'LotArea', 'LotConfig_CulDSac', 'LowQualFinSF', 'Neighborhood_BrkSide', 'Neighborhood_Crawfor', 'Neighborhood_NWAmes', 'Neighborhood_NoRidge', 'Neighborhood_NridgHt', 'Neighborhood_Somerst', 'Neighborhood_StoneBr', 'OverallCond', 'OverallQual', 'PoolArea', 'RoofMatl_WdShngl', 'SaleCondition_Normal', 'SaleType_New', 'TotalBsmtSF', 'WoodDeckSF', 'YearBuilt']上下滑動查看更多遺傳算法-優化歷史上面是一段優化算法的描述,該算法用于在1000代進化過程中尋找最佳特征組合。進化過程將特征的受歡迎程度用熱圖可視化表示,橫向代表不同特征,縱向代表進化代數。熱圖上顏色越深,代表該特征在該代群體中越受歡迎。從熱圖可以看出,一些特征在整個進化過程中保持較高受歡迎度;另一些特征則很快被淘汰;還有些特征的受歡迎程度隨著進化代數的增加而有所波動。該可視化展示了進化算法在特征選擇過程中的動態變化,有助于了解算法的工作原理和特征的重要性。
各種方法之間的比較
我們嘗試了三種不同的技術:SFS、CMA-ES 和 GA。就找到的最佳目標和找到目標所花費的時間而言,它們之間的比較如何?
這些測試是在一臺安裝了Ubuntu 22.04和Python 3.11.7的AMD Ryzen 7 5800X3D(8/16核)機器上進行的。SFS和GA分別通過一個包含16個Worker的多進程池來運行目標函數。CMA-ES是單進程的,多進程運行似乎沒有明顯的改進,但我相信如果有更多的工作來使算法并行化,情況會有所改善。
這些是執行時間。對SFS來說,是總執行時間。對于CMA-ES和GA,這是達到最佳解決方案所需的時間。時間越短越好。
SFS: 42.448 sec
GA: 158.027 sec
CMA-ES: 48.326 sec目標函數被調用的次數--越少越好:
SFS: 22791
GA: 600525
CMA-ES: 20000為目標函數找到的最佳值,與基準值相比——越少越好:
baseline BIC: 34570.1662
SFS: 33708.9860
GA: 33705.5696
CMA-ES: 33703.0705在目標函數方面,遺傳算法(GA)能夠取得比序列前向選擇(SFS)更好的結果,因為它能夠利用盡可能多的CPU內核來并行運行目標函數,但代價是它的運行速度是所有算法中最慢的。GA調用目標函數的次數遠遠超過其他方法。對超參數進行進一步優化可能會提高GA的性能。
相比之下,SFS的運行速度很快(能夠在所有CPU內核上并行運行),但其性能一般。SFS是所有算法中最簡單的。
如果你只是想快速估算出近似最優的特征集,而不太在意精度,SFS是一個不錯的選擇。
然而,如果你追求最優目標值,無約束進化策略(CMA-ES)似乎是最好的選擇。